Reconnaissance de chiffres manuscrits sur Python
 Image credit: Josef Steppan
Image credit: Josef Steppan
1. Données MNIST
Nous commençons d’abord par importer les librairies nécessaires.
import numpy as np
import pandas as pd
from matplotlib import pyplot as plt
import random
import time
from keras.datasets import mnist
La base de données se télécharge facilement avec le package keras comme suit :
(X0, Y0), (X1, Y1) = mnist.load_data()
$X_0(i, :, :)$ est la $i^{ème}$ image de la base d’apprentissage ; elle est de taille $28 \times 28$. On peut l’afficher avec la fonction imshow de matplotlib.pyplot. Les variables $Y_0$ et $Y_1$ contiennent les classes des observations dans chacune des bases. Les classes sont étiquetées de $0$ à $9$.
Avant toute analyse, on va préparer les données de la manière suivante :
# On met le type des images en float.
X0 = X0.astype('float32')
X1 = X1.astype('float32')
# On met les images sous la forme d'un vecteur.
X0 = X0.reshape(60000, 784)
X1 = X1.reshape(10000, 784)
# On normalise les images.
X0 = X0 / 255.0
X1 = X1 / 255.0
Nous pouvons alors afficher l’une des images.
#Affichage de la 3ème image
n = 2
Y0_ = pd.get_dummies(Y0).values
Y1_ = pd.get_dummies(Y1).values
plt.imshow(X0[n, :].reshape(28, 28), cmap="gray")
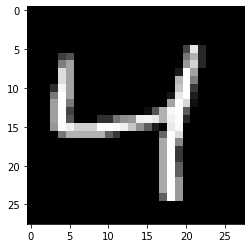
2. Régression multinomiale
\begin{equation} \label{eqn:softmax_proba} \forall k \in {1,\cdots, K}, \mathbb{P}(Y = k) = \frac{\exp(\langle \theta^{(k)}, \tilde{x} \rangle )}{\sum_{m=1}^K \exp(\langle \theta^{(m)}, \tilde{x} \rangle )},\mathrm{avec} \tilde{x} = \left( \begin{array}{c} 1 \ x \end{array} \right). \end{equation}
Les estimateurs du maximum de vraisemblance de l’échantillon sont les paramètres $\theta$ qui minimisent le critère d’entropie croisée: \begin{equation} \label{eqn:entropiecroisee} \mathcal{S}(\theta) = - \frac{1}{n} \sum_{i=1}^n \sum_{k=1}^K z_i^{(k)} \log\left(p_i^{(k)}(\theta) \right),\end{equation} avec \begin{equation} z_i^{(k)} = \mathbf{1}_{y_i = k} \mathrm{et} p_i^{(k)}(\theta) = \frac{\exp(\langle \theta^{(k)}, \tilde{x}_i \rangle )}{\sum_{m=1}^K \exp(\langle \theta^{(m)}, \tilde{x}_i \rangle )}. \end{equation}
3. Descente de gradient
\begin{equation} \label{eqn:miseajour} \theta \longleftarrow \theta - \rho \nabla_{\theta} \mathcal{S}(\theta), \end{equation} où $\rho>0$ désigne un pas fixe et $\nabla_{\theta} \mathcal{S}$ le gradient de $\mathcal{S}$ par rapport à $\theta$.
Le gradient de l’entropie croisée $\mathcal{S}$ par rapport à $\theta^{(k)}$ en $\theta$ vaut : $$ \nabla_{\theta^{(k)}} \mathcal{S}(\theta) = \frac{1}{n} \sum_{i=1}^{n} \left(p_i^{(k)}(\theta) - z_i^{(k)}\right) \tilde{x}_i . $$
3. Application de l’algorithme de descente de gradient sur les données MNIST
def RegMultinomialGradDesc(X_train, Y_train, X_test, Y_test, Y_train_list, Y_test_list, epochs, alpha):
"""
Calcul du gradient, theta et entropie croisée
Paramétres
----------
X_train : ndarray (n x p)
Y_train : ndarray (n x K)
epochs : nombre d'epoques à réaliser
alpha : scalaire
X_test : ndarray (m x p)
Y_test : ndarray (m x K)
Returns
-------
beta : ndarray (p+1 x K)
Lvals_train : liste contenant les valeurs de l'entropie sur les données de train à toutes les epoques
Lvals_test : liste contenant les valeurs de l'entropie sur les données de validation à toutes les epoques
Learning_T : liste contenant les temps d'exécution des époques
scores_train : liste contenant les scores sur les données de train à toutes les époques
scores_test : liste contenant les scores sur les données de validation à toutes les époques
"""
Epochs = epochs
N, p = X_train.shape
X_train = np.insert(X_train, 0, 1, axis=1) # on insère des 1 à la position 0 (colonne 0) de X
X_test = np.insert(X_test, 0, 1, axis=1) # on insère des 1 à la position 0 (colonne 0) de X
K = Y_train.shape[1]
Theta = np.zeros((p+1, K))
Z_train = X_train[:, 1::]
Z_test = X_test[:, 1::]
Learning_T = []
Lvals_train = []
Lvals_test = []
scores_train = []
scores_test = []
for epoch in range(Epochs):
start_time = time.time()
L_train = eval_L(X_train, Y_train, Theta)
L_test = eval_L(X_test, Y_test, Theta)
Lvals_train.append(L_train)
Lvals_test.append(L_test)
print("Epoch " + str(epoch) + " : " + " Cost train est : " + str(L_train) + " et" + " Cost test est : " + str(L_test))
Theta = Theta - (1/N)*alpha*Gradient(X_train, Y_train, Theta)
prediction_Xtrain = prediction_Label(Z_train, Theta)
numClassCorrect_train = 0
for i in range(N):
if prediction_Xtrain[i]==Y_train_list[i]:
numClassCorrect_train+=1
accuracy_train = numClassCorrect_train/N
scores_train.append(accuracy_train)
prediction_Xtest = prediction_Label(Z_test, Theta)
N_test = Z_test.shape[0]
numClassCorrect_test = 0
for i in range(N_test):
if prediction_Xtest[i]==Y_test_list[i]:
numClassCorrect_test+=1
accuracy_test = numClassCorrect_test/N_test
scores_test.append(accuracy_test)
learning_time = time.time() - start_time
Learning_T.append(learning_time)
return Theta, Learning_T, Lvals_train, Lvals_test, scores_train, scores_test
alpha = 0.01 # fixation du pas
epochs = 100 # fixation du nombre d'epochs
X_train = X0[:50000]
Y_train = Y0_[:50000] # Y sous forme d'une matrice n, K
X_test = X1
Y_test = Y1_
Y_train_list = Y0[:50000] # Y sous forme d'une liste de longueur n
Y_test_list = Y1
Theta, Learning_T, entrop_train, entrop_test, score_train, score_test = RegMultinomial_GradDesc(X_train, Y_train, X_test, Y_test, Y_train_list, Y_test_list, epochs, alpha)
Appliquons l’algorithme de descente de gradient sur les données MNIST en prenant 50000 données d’apprentissage et 10000 de validation.
alpha = 0.01 # fixation du pas
epochs = 100 # fixation du nombre d'epochs
X_train = X0[:50000]
Y_train = Y0_[:50000] # Y sous forme d'une matrice n, K
X_test = X1
Y_test = Y1_
Y_train_list = Y0[:50000] # Y sous forme d'une liste de longueur n
Y_test_list = Y1
Theta, Learning_T, entrop_train, entrop_test, score_train, score_test = RegMultinomial_GradDesc(X_train, Y_train, X_test, Y_test, Y_train_list, Y_test_list, epochs, alpha)
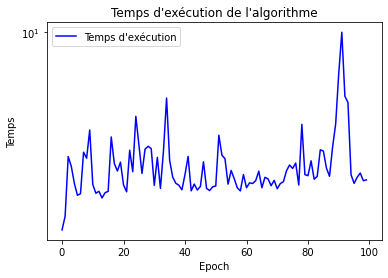
epoch = range(epochs)
plt.figure("")
plt.title("Temps d'exécution de l'algorithme")
plt.xlabel("Epoch")
plt.ylabel("Temps")
plt.yscale('symlog')
plt.plot(epoch, Learning_T, label = "Temps d'exécution", color='blue')
plt.legend()
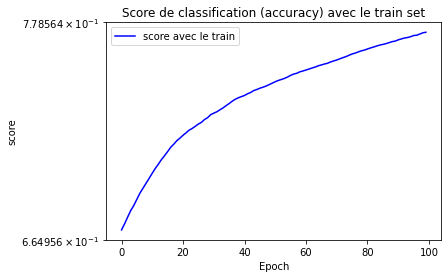
epoch = range(epochs)
plt.figure("")
plt.title("Score de classification (accuracy) avec le train set")
plt.xlabel("Epoch")
plt.ylabel("score")
plt.yscale('symlog')
plt.plot(epoch, score_train, label = "score avec le train", color='blue')
plt.legend()
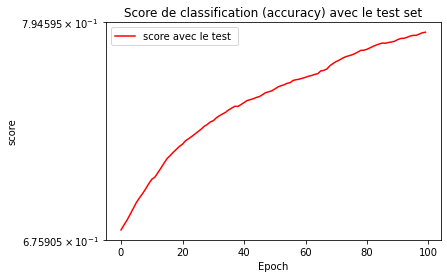
epoch = range(epochs)
plt.figure("")
plt.title("Score de classification (accuracy) avec le test set")
plt.xlabel("Epoch")
plt.ylabel("score")
plt.yscale('symlog')
plt.plot(epoch, score_test, label = "score avec le test ", color='red')
plt.legend()
4. Descente de gradient stochastique par mini-lots
Modifions de l'algorithme précédent afin d'inclure des mises à jour sur mini-lots et appliquons le aux données MNIST.
Définissons d’abord la fonction qui calcule le gradient par mini-lots.
def Gradient_miniLot(X, Y, Theta, m):
N, p = X.shape
K = Y.shape[1]
#m = 256
Gradient = np.zeros((p, K))
T = range(N)
random.seed(10)
Bj = random.sample(T, int(m))
for i in Bj:
XiHat = X[i]
XiHat = XiHat.reshape(1, XiHat.shape[0])
Yi = Y[i]
Yi = Yi.reshape(1, Yi.shape[0])
qi = Soft_Max(XiHat, Theta)
Gradient += np.outer(XiHat, qi - Yi)
return Gradient
Nous pouvons maintenant définir celle qui inclut les mini-lots.
def RegMultinomial_GradDescMinibatch(X_train, Y_train, X_test, Y_test, Y_train_list, Y_test_list, alpha, m):
"""
Calcul du gradient, theta et entropie croisée
Parameters
----------
X_train : ndarray (n x p)
Y_train : ndarray (n x K)
epochs : nombre d'epoques à réaliser
alpha : scalaire
m : nombre de lots
X_test : ndarray (m x p)
Y_test : ndarray (m x K)
Returns
-------
beta : ndarray (p+1 x K)
Lvals_train : liste contenant les valeurs de l'entropie sur les données de train à toutes les epoques
Lvals_test : liste contenant les valeurs de l'entropie sur les données de validation à toutes les epoques
Learning_T : liste contenant les temps d'exécution des époques
scores_train : liste contenant les scores sur les données de train à toutes les époques
scores_test : liste contenant les scores sur les données de validation à toutes les époques
"""
N, p = X_train.shape
Epochs = int(N/m)
X_train = np.insert(X_train, 0, 1, axis=1) # on insère des 1 à la position 0 (colonne 0) de X
X_test = np.insert(X_test, 0, 1, axis=1) # on insère des 1 à la position 0 (colonne 0) de X
K = Y_train.shape[1]
Theta = np.zeros((p+1, K))
Z_train = X_train[:, 1::]
Z_test = X_test[:, 1::]
Learning_T = []
Lvals_train = []
Lvals_test = []
scores_train = []
scores_test = []
for epoch in range(Epochs):
start_time = time.time()
L_train = eval_L_minibatch(X_train, Y_train, Theta, m)
L_test = eval_L_minibatch(X_test, Y_test, Theta, m)
Lvals_train.append(L_train)
Lvals_test.append(L_test)
print("Epoch " + str(epoch) + " : " + " Cost train est : " + str(L_train) + " et" + " Cost test est : " + str(L_test))
Theta = Theta - (1/N)*alpha*Gradient_miniLot(X_train, Y_train, Theta, m)
prediction_Xtrain = prediction_Label(Z_train, Theta)
numClassCorrect_train = 0
for i in range(N):
if prediction_Xtrain[i]==Y_train_list[i]:
numClassCorrect_train+=1
accuracy_train = numClassCorrect_train/N
scores_train.append(accuracy_train)
prediction_Xtest = prediction_Label(Z_test, Theta)
N_test = Z_test.shape[0]
numClassCorrect_test = 0
for i in range(N_test):
if prediction_Xtest[i]==Y_test_list[i]:
numClassCorrect_test+=1
accuracy_test = numClassCorrect_test/N_test
scores_test.append(accuracy_test)
learning_time = time.time() - start_time
Learning_T.append(learning_time)
return Theta, Learning_T, Lvals_train, Lvals_test, scores_train, scores_test
Application de l’algorithme de descente de gradient stochastique par mini-lots sur les données MNIST en prenant 50000 données d’apprentissage et 10000 de validation.
alpha = 0.01 # fixation du pas
m = 256 # fixation du nombre d'epochs
X_train = X0[:50000]
Y_train = Y0_[:50000] # Y sous forme d'une matrice n, K
X_test = X1
Y_test = Y1_
Y_train_list = Y0[:50000] # Y sous forme d'une liste de longueur n
Y_test_list = Y1
Theta, Learning_T, entrop_train, entrop_test, score_train, score_test = RegMultinomial_GradDescMinibatch(X_train, Y_train, X_test, Y_test, Y_train_list, Y_test_list, alpha, m)
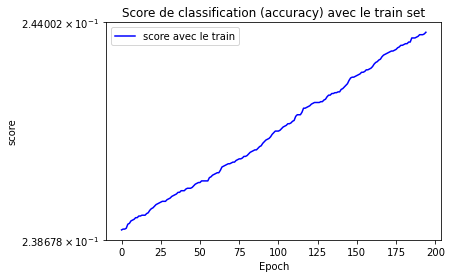
plt.figure("")
plt.title("Score de classification (accuracy) avec le train set")
plt.xlabel("Epoch")
plt.ylabel("score")
plt.yscale('symlog')
plt.plot(epoch, score_train, label = "score avec le train", color='blue')
plt.legend()
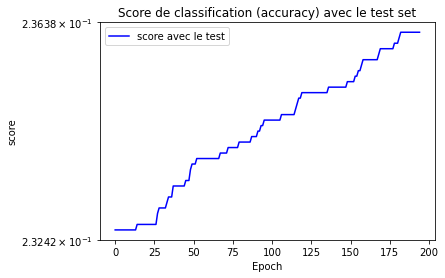
plt.figure("")
plt.title("Score de classification (accuracy) avec le test set")
plt.xlabel("Epoch")
plt.ylabel("score")
plt.yscale('symlog')
plt.plot(epoch, score_test, label = "score avec le test", color='blue')
plt.legend()
5. Régression multinomiale en keras
Ici, nous allons mettre en oeuvre en keras le modèle de régression multinomiale.
# Importation des librairies nécessaires
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import tensorflow as tf
from tensorflow import keras
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense, Activation
from keras.regularizers import l2
# Training set et test set
(X0, Y0), (X1, Y1) = tf.keras.datasets.mnist.load_data()
# On met les images sous la forme de vecteur.
X0 = X0.reshape(60000, 784)
X1 = X1.reshape(10000, 784)
# On normalise les images.
X0 = X0 / 255.0
X1 = X1 / 255.0
# Transformation de Y0 en matrice N X K avec N le nompbre d'observations et K le nombre de classes
Y0_ = pd.get_dummies(Y0).values
# Constitution d'un ensemble d'entrainement composé de 50000 observations
# et d'un ensemble test composé de 10000 observations
X_train = X0[:50000]
Y_train = Y0_[:50000]
X_valid = X0[:20000]
Y_valid = Y0[:20000]
X_test = X1
Y_test = Y1
Nous pouvons dès à présent construire le modèle de régression multinomiale sous keras.
# Variables et classes du modèle
input_dim = X_train.shape[1] # 784 variables
output_dim = Y0_.shape[1] # 10 classes
# Construction du modèle de régression multinomiale
def classification_model():
model = Sequential()
model.add(Dense(output_dim, input_dim=input_dim, activation='softmax'))
model.compile(loss='categorical_crossentropy', optimizer='sgd', metrics=['accuracy'])
return model
# Entrainement du modèle
keras_model = classification_model()
keras_model.fit(X_train, Y_train, epochs=300, verbose=0)
Qu’en est-il de la performance du modèle sur les données de validation?
# Calcul du degré de précision ou accuracy sur X_valid
classes = np.argmax(keras_model.predict(X_valid), axis = 1)
N_valid = X_valid.shape[0]
numClassCorrect = 0
for i in range(N_valid):
if classes[i]==Y_valid[i]:
numClassCorrect+=1
accuracy = numClassCorrect/N_valid
print('Accuracy sur X_valid : ' + str(accuracy))
Accuracy sur X_valid : 0.9348
Allons voir la précision sur les données test.
# Prédictions de X_test
classes = np.argmax(keras_model.predict(X_test), axis = 1)
classes.shape
(10000,)
# Calcul du degré de précision ou accuracy sur X_test
N_test = X_test.shape[0]
numClassCorrect = 0
for i in range(N_test):
if classes[i]==Y_test[i]:
numClassCorrect+=1
accuracy = numClassCorrect/N_test
print('Accuracy sur X_test : ' + str(accuracy))
Accuracy sur X_test : 0.9254
X_train = X0[:1000]
Y_train = Y0_[:1000]
X_valid = X0[:400]
Y_valid = Y0[:400]
# Entrainement du modèle
keras_model = classification_model()
keras_model.fit(X_train, Y_train, epochs=300, verbose=0)
# Calcul du degré de précision ou accuracy sur X_valid
classes = np.argmax(keras_model.predict(X_valid), axis = 1)
N_valid = X_valid.shape[0]
numClassCorrect = 0
for i in range(N_valid):
if classes[i]==Y_valid[i]:
numClassCorrect+=1
accuracy = numClassCorrect/N_valid
print('Accuracy sur X_valid : ' + str(accuracy))
Accuracy sur X_valid : 0.9675
# Prédictions de X_test
classes = np.argmax(keras_model.predict(X_test), axis = 1)
classes.shape
(10000,)
# Calcul du degré de précision ou accuracy sur X_test
N_test = X_test.shape[0]
numClassCorrect = 0
for i in range(N_test):
if classes[i]==Y_test[i]:
numClassCorrect+=1
accuracy = numClassCorrect/N_test
print('Accuracy sur X_test : ' + str(accuracy))
Accuracy sur X_test : 0.8662
accuracy_mnist = []
for i in range(1, 31):
# Entrainement du modèle
keras_model = classification_model()
keras_model.fit(X_train, Y_train, epochs=i, verbose=0)
# Prédictions de X_test
classes = np.argmax(keras_model.predict(X_test), axis = 1)
classes.shape
# Calcul du degré de précision ou accuracy sur X_test
N_test = X_test.shape[0]
numClassCorrect = 0
for i in range(N_test):
if classes[i]==Y_test[i]:
numClassCorrect+=1
accuracy = numClassCorrect/N_test
accuracy_mnist.append(accuracy)
epoque = range(1, 31)
plt.title("Score de classification en fonction du nombre d'époque")
plt.xlabel("Epoch")
plt.ylabel("Score")
plt.yscale('symlog')
plt.plot(epoque, accuracy_mnist)
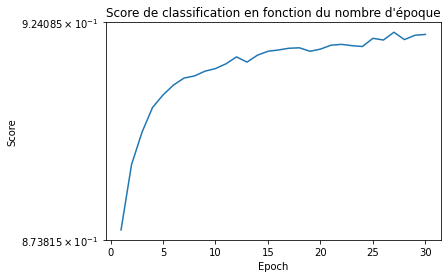
print(accuracy_mnist.index(max(accuracy_mnist)))
26
| Type | Cartes | Noyau | Pas | Activation |
|---|---|---|---|---|
| Conv. | 6 | 5x5 | 1 | tanh |
| Av. Pooling | 6 | 2x2 | 2 | - |
| Conv. | 12 | 5x5 | 1 | tanh |
| Av. Pooling | 12 | 2x2 | 4 | - |
| Dense | - | 10 | - | softmax |
Dimension des images
Nombre de paramètres à chaque couche
Comparaison avec le modèle dense de la régression multinomiale
Application du modèle aux données MNIST et comparaison aux résultats obtenus avec la régression multinomiale.
# Importation des librairies nécessaires
import numpy as np
import mnist
from tensorflow import keras
# Images de train et de test
train_img = mnist.train_images()
train_label = mnist.train_labels()
test_img = mnist.test_images()
test_label = mnist.test_labels()
# Affichage de la dimension des images
print(train_img.shape) ### (60000, 28, 28)
print(train_label.shape) ### (60000,)
print(test_img.shape) ### (60000, 28, 28)
print(test_label.shape) ### (60000,)
(60000, 28, 28)
(60000,)
(10000, 28, 28)
(10000,)
# Normalisation des images. La raison de la normalisation est de s'assurer que l'ensemble
# constitué d'images a une moyenne de 0 et un écart type de 1 (ici nous allons passer de [0, 255] à [-0.5, 0.5]).
#Les avantages de cela se voient dans la réduction du temps d'apprentissage.
train_img = (train_img / 255) - 0.5
test_img = (test_img / 255) - 0.5
# Reshape des images (car keras exige la troisièeme dimension)
train_img = np.expand_dims(train_img, axis=3)
test_img = np.expand_dims(test_img, axis=3)
print(train_img.shape) ### (60000, 28, 28, 1)
print(test_img.shape) #### (10000, 28, 28, 1)
(60000, 28, 28, 1)
(10000, 28, 28, 1)
valid_img = train_img[:5000]
valid_label = train_label[:5000]
lenet_5_model = keras.models.Sequential([
keras.layers.Conv2D(6, kernel_size=5, strides=1, activation='tanh', input_shape=train_img[0].shape, padding='same'),
keras .layers.AveragePooling2D(),
keras.layers.Conv2D(16, kernel_size=5, strides=1, activation='tanh', padding='valid'),
keras.layers.AveragePooling2D(),
keras.layers.Flatten(),
keras.layers.Dense(120, activation='tanh'),
keras.layers.Dense(84, activation='tanh'),
keras.layers.Dense( 10, activation='softmax')
])
Compilation et construction du modèle
# Ici, nous compilons le modèle dèjà implémenté en arrière-plan en fournissant des caractéristiques
# supllémentaires : fonction de perte, optimiseur et la métrique
lenet_5_model.compile(optimizer='adam', loss=keras.losses.sparse_categorical_crossentropy, metrics=['accuracy'])
# Nous entrainons le modèle en utilisant une fonction de perte (différence entre valeurs prédites
# par le réseau et les valeurs réelles de l'ensemble d'entrainement), un algorithme d'optimisation
# (adam) afin de faciliter le nombre de modifications apportées aux poids au sein du réseau, un
# ensemble de validation à chaque époque.
lenet_5_model.fit(train_img, train_label, epochs=5, validation_data=(valid_img, valid_label))
# Evaluation sur le jeu de données test
lenet_5_model.evaluate(test_img, test_label)
313/313 [==============================] - 1s 4ms/step - loss: 0.0612 - accuracy: 0.9808
[0.06115240976214409, 0.9807999730110168]
L'analyse des résultats obtenus à l'issue de cette évaluation montre une précision de 98%, ce qui est très intéressant.
Voici quelques caractéristiques du modèle par couche.
Model: "sequential_1"
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
conv2d (Conv2D) (None, 28, 28, 6) 156
average_pooling2d (AverageP (None, 14, 14, 6) 0
ooling2D)
conv2d_1 (Conv2D) (None, 10, 10, 16) 2416
average_pooling2d_1 (Averag (None, 5, 5, 16) 0
ePooling2D)
flatten (Flatten) (None, 400) 0
dense_1 (Dense) (None, 120) 48120
dense_2 (Dense) (None, 84) 10164
dense_3 (Dense) (None, 10) 850
=================================================================
Total params: 61,706
Trainable params: 61,706
Non-trainable params: 0
_________________________________________________________________
Apprentissage du réseau sur 6000 exemples et comparaison des performances avec le réseau dense appris dans les mêmes conditions.
train_img2 = train_img[:6000]
train_label2 = train_label[:6000]
valid_img2 = train_img2[:500]
valid_label2 = train_label2[:500]
Entrainement du modèle
#Entrainement du modèle sur le jeu de données réduit
lenet_5_model.fit(train_img2, train_label2, epochs=5, validation_data=(valid_img2, valid_label2))
# Evaluation sur le jeu de données test
lenet_5_model.evaluate(test_img, test_label)
313/313 [==============================] - 1s 4ms/step - loss: 0.0533 - accuracy: 0.9853
[0.053335435688495636, 0.9853000044822693]
Outil utilisé : Python
License
Copyright 2020-present Mamoudou KOUME.